本数据集包含不同类型的脑部 MRI 影像样本:脑胶质瘤(Glioma)、脑膜瘤(Meningioma)、垂体瘤(Pituitary Tumor) 以及 正常脑部(Normal)。数据集旨在构建高精度的图像分类模型,实现对脑肿瘤与健康脑组织的自动识别与判定。通过对大量标注清晰、质量可控的MRI影像进行深度学习训练,模型能够自动提取病灶的关键结构与纹理特征,区分不同肿瘤类型并输出类别标签与置信度评分,从而为临床提供可靠的辅助诊断依据。该数据集的建立旨在推动深度学习与人工智能技术在脑肿瘤早期筛查、术前评估与智能诊断中的应用,助力实现脑部医学影像分析的标准化与智能化。
数据集信息
编号:Dataset-17
大小:86M
整理:Bob
数据集概览
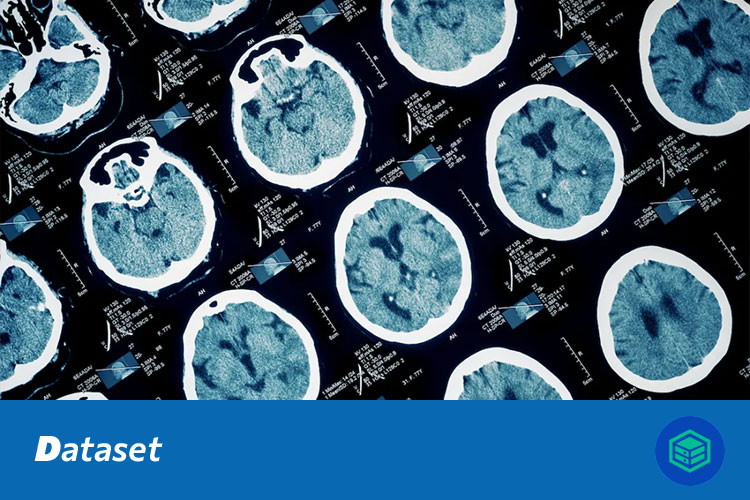
数据图像:
图1 样本图像
数据类型:

表1 数据类型与格式
数据规模:
(1)数据集划分饼图
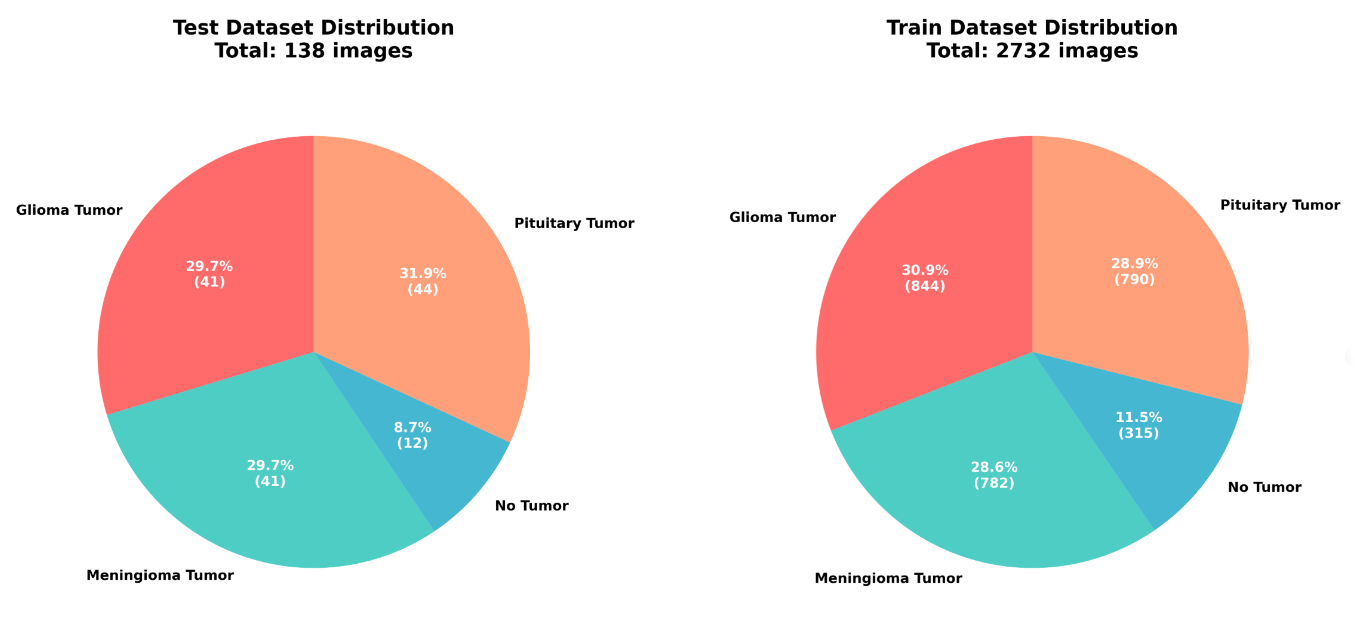
图2 数据集划分饼图
数据集类别

表3 类别定义
数据集来源

表4 数据集来源与说明
数据集用途
本数据集用于脑肿瘤 MRI 图像的分类模型训练与验证,包含四类样本:脑胶质瘤(Glioma)、脑膜瘤(Meningioma)、垂体瘤(Pituitary Tumor)以及正常脑部(Normal)。可应用于以下研究与开发方向:
(1)模型训练:支持基于深度学习的图像分类模型(如 ResNet50、VGG16、Swin Transformer)进行脑部 MRI 影像特征学习。
(2)性能评估:用于测试模型在多类别脑肿瘤分类任务中的准确率、召回率、F1 值等指标。
(3)特征分析:研究不同肿瘤类型与正常脑组织在结构、纹理与信号强度等方面的差异。
(4)系统开发:为脑肿瘤智能诊断系统、医学影像辅助诊断平台及筛查系统提供数据支持。
数据集须知
(1)数据来源:基于公开的脑肿瘤 MRI 影像数据集(如 Kaggle 等),仅限科研与教学用途。
(2)数据结构:包含训练集与测试集,分为四类样本(脑胶质瘤、脑膜瘤、垂体瘤、正常脑部)。
(3)文件格式:图像文件为 .jpg 格式(按类别文件夹组织),文件命名与类别标签严格对应。
(4)使用要求:需遵守医学影像数据隐私与伦理保护相关规定,引用时须注明数据来源。
(5)适用范围:适用于脑肿瘤 MRI 影像的识别、分类、特征提取及智能诊断系统研究。
数据集性能
训练与验证准确率和损失曲线:
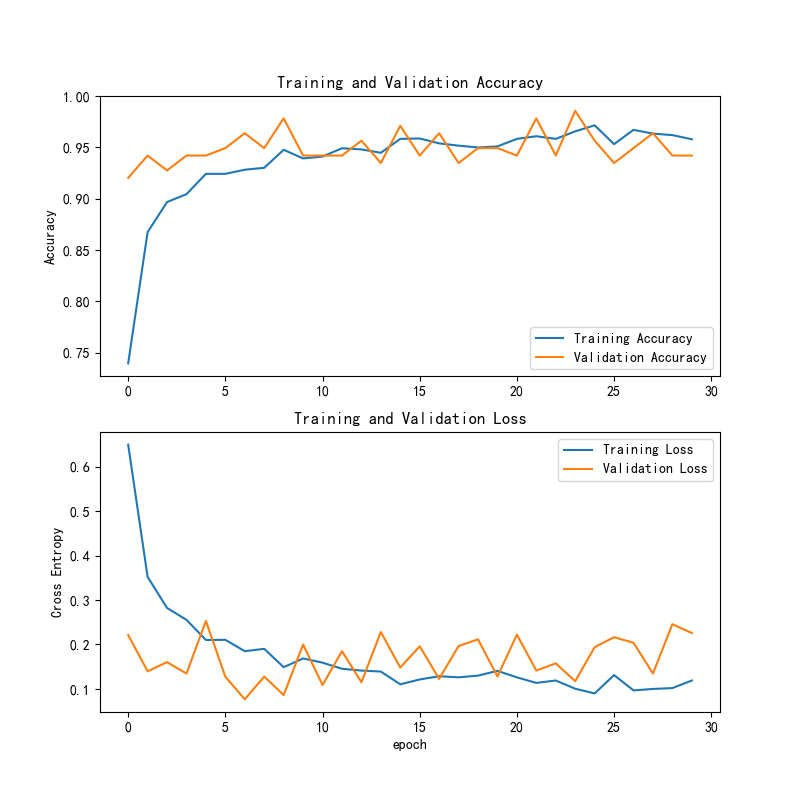
图3 训练与验证准确率和损失曲线线
前10个epoch准确率快速上升后稳定在0.94–0.97区间,训练与验证曲线贴合,未见明显过拟合;训练损失持续下降,验证损失小幅波动,整体收敛良好。若要进一步稳固泛化,可增加正常样本多样性与难例增强,并尝试阈值校准以降低正常类误报。
混淆矩阵热力图:
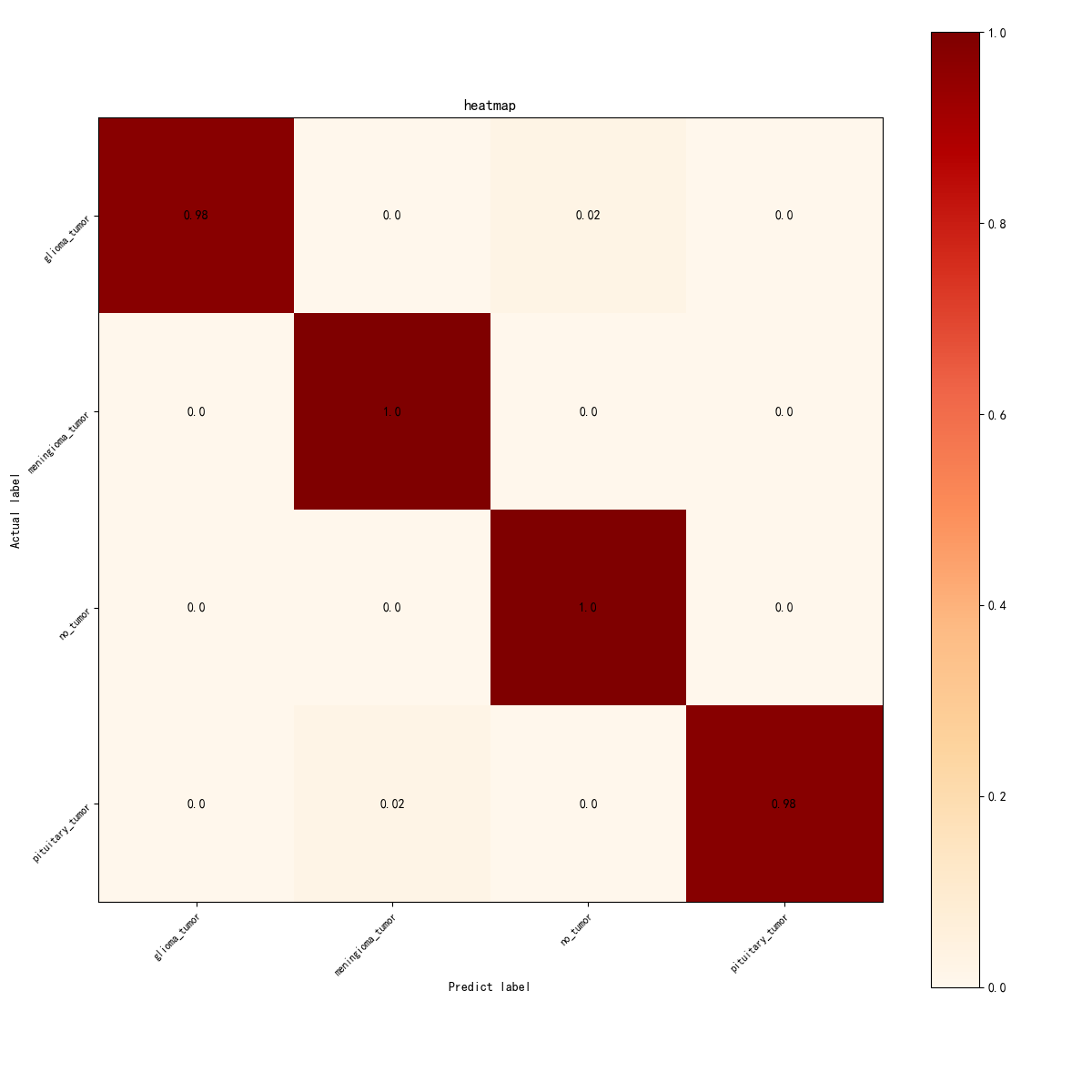
图4 混淆矩阵热力图
预测基本集中在对角线,整体分类清晰;少量误分主要为 glioma → no_tumor(约2%) 与 pituitary → meningioma(约2%),符合这两类在某些切片上影像表征相似、边界不清的临床特点;其余类别几乎无相互混淆,体现模型对关键特征的良好区分能力。
各类的分类性能评估:准确率、精确率、召回率与F1分数图

图5 各类认知障碍的分类性能评估:准确率、精确率、召回率与F1分数图
模型总体准确率约0.99,各类F1均在0.96–0.99之间,glioma、meningioma、pituitary 的Precision/Recall均接近满分;唯独 no_tumor 的Precision偏低(≈0.92),提示正常样本存在一定误报,但其Recall为1.00,说明对正常的漏检极少,整体表现稳健、均衡。
联系我们

官方声明
(1)实验环境真实性与合规性声明:
本研究所使用的硬件与软件环境均为真实可复现的配置,未采用虚构实验平台或虚拟模拟环境。实验平台为作者自主购买的惠普(HP)暗影精灵 10 台式整机,具体硬件参数详见表。软件环境涵盖操作系统、开发工具、深度学习框架等,具体配置详见表,所有软件组件均来源于官方渠道或开源社区,并按照其许可协议合法安装与使用。
研究过程中严格遵循学术诚信和实验可复现性要求,确保所有实验数据、训练过程与结果均可在相同环境下被重复验证,符合科研规范与工程实践标准。
(2)版权声明:
本算法改进中涉及的文字、图片、表格、程序代码及实验数据,除特别注明外,均由2zcode.Bob独立完成。未经2zcode官方书面许可,任何单位或个人不得擅自复制、传播、修改、转发或用于商业用途。如需引用本研究内容,请遵循学术规范,注明出处,并不得歪曲或误用相关结论。
本研究所使用的第三方开源工具、框架及数据资源均已在文中明确标注,并严格遵守其相应的开源许可协议。使用过程中无违反知识产权相关法规,且全部用于非商业性学术研究用途。


评论(0)